AI赋能蛋白质研究:SaprotHub让蛋白质AI模型训练和调用不再有门槛!
来源:机器之心
时间:2025-01-23 23:24:39 428浏览 收藏
学习知识要善于思考,思考,再思考!今天golang学习网小编就给大家带来《AI赋能蛋白质研究:SaprotHub让蛋白质AI模型训练和调用不再有门槛!》,以下内容主要包含等知识点,如果你正在学习或准备学习科技周边,就都不要错过本文啦~让我们一起来看看吧,能帮助到你就更好了!

编辑 | ScienceAI
近年来,AI 技术在蛋白质研究领域发挥了越来越重要的作用。从 AlphaFold2 在结构预测任务上的脱颖而出,到各类蛋白质语言模型(PLMs)在功能预测方面的重大进展,生物研究者们可以利用各式各样的 AI 模型来辅助他们的研究。
然而,随着模型变得越来越复杂,如何训练和调用这些 AI 模型对于缺乏机器学习专业知识的研究者来说是一件非常具有挑战的任务,也因此限制了 AI 技术在实际研究中的应用。
为了解决这个限制,来自西湖大学的研究人员推出了 SaprotHub,允许生物学家以交互的方式训练和调用 AI 模型,同时提出了社区共建的概念,鼓励用户分享训练好的模型权重,促进领域的生态发展。
该研究以「SaprotHub: Making Protein Modeling Accessible to All Biologists」为题,于 2024 年 12 月 13 日更新在 bioRxiv 预印本平台。

SaprotHub 平台
SaprotHub 包括三个核心部分:Saprot 模型、ColabSaprot 交互界面以及 HuggingFace 模型仓库。用户通过 ColabSaprot 来训练和使用 Saprot 模型,同时可以直接加载 HuggingFace 仓库中的模型来进行预测。
关于 Saprot 的详细介绍,请参考作者的文章分享《基于结构感知词表的蛋白质语言模型 SaProt》。
文章链接:https://zhuanlan.zhihu.com/p/664754366
ColabSaprot
ColabSaprot 以交互界面的形式让用户可以不需要编写任何代码就能够训练和调用模型,包括一键配置环境、自动处理数据、模型训练监控、最佳权重保存等。
ColabSaprot 目前提供三大功能:模型训练、模型调用以及模型分享。详细功能介绍如下:
模型训练
ColabSaprot 将训练模型所需的繁杂步骤全部进行了封装。用户只需要在交互界面中按照需求设定好目标训练任务,随后上传一个指定格式的数据集文件即可开始训练。
考虑到使用者可能对相关的机器学习知识并不熟悉(例如 learning rate、batch size 等),ColabSaprot 在每一个选择栏都有相关的提示说明,确保使用者能够轻松地使用 ColabSaprot 完成自己的模型训练。
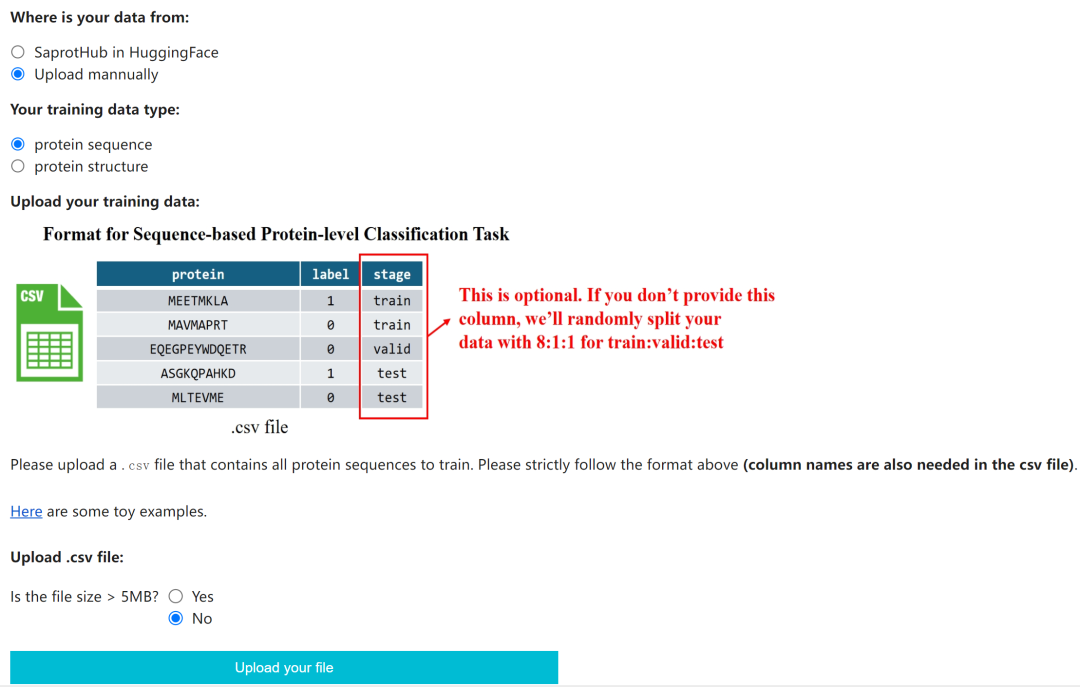
图示:ColabSaprot 模型训练界面。(来源:ColabSaprot 平台截图)
模型调用
ColabSaprot 提供了多种预测任务供用户调用,包括蛋白质属性预测(利用自己训练好的模型或者 SaprotHub 模型仓库的模型进行预测)、突变效应预测(预测单点突变或组合突变对蛋白质 fitness 的影响,例如酶活性等)、蛋白质序列设计(根据上传的蛋白质骨架反向设计序列)以及蛋白质表征获取(获取模型生成的蛋白质 embedding 进行后续分析)。
用户可以自由选择想要使用的功能,跟随指引即可轻松使用。
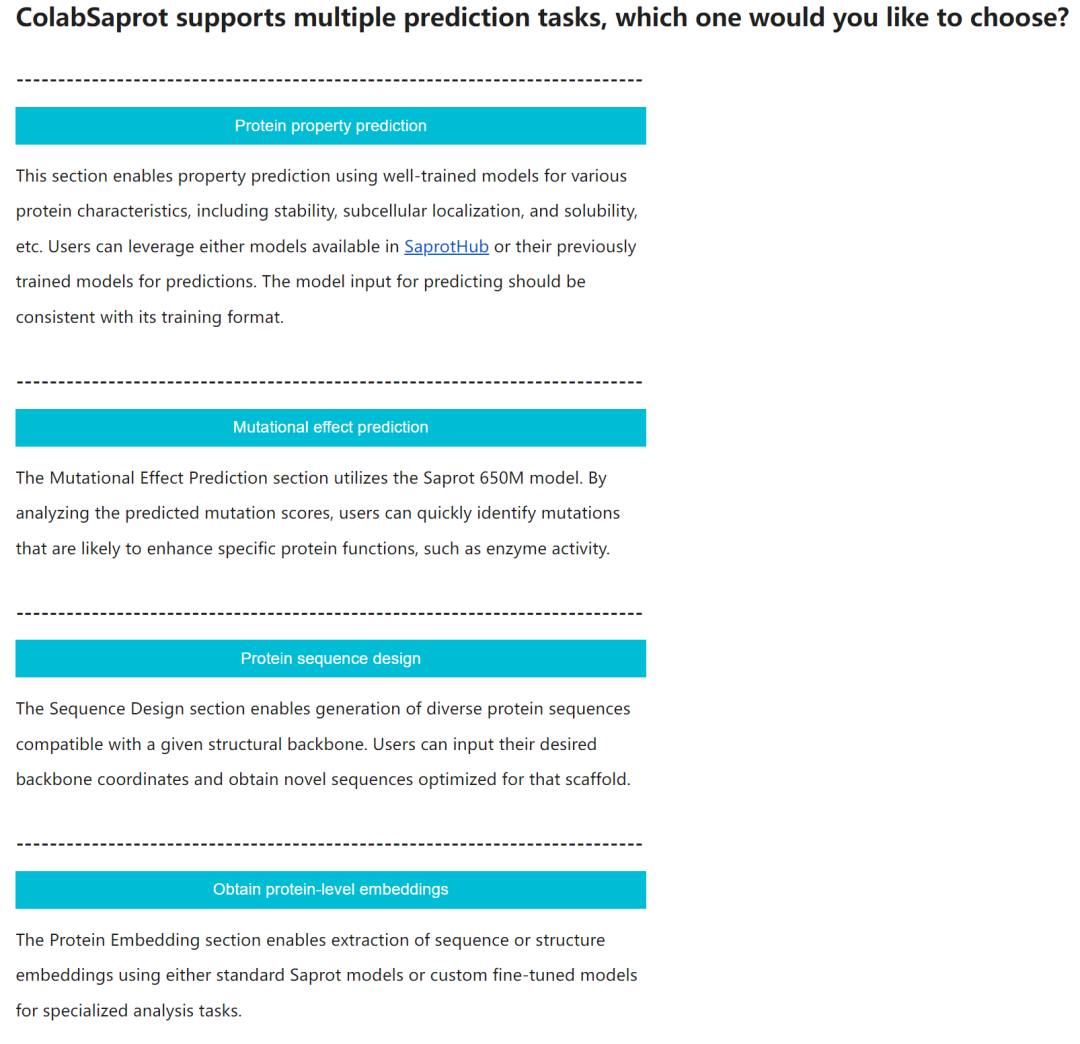
图示:ColabSaprot 支持多种预测任务。(来源:ColabSaprot 平台截图)
模型分享
为了鼓励用户分享自己训练的模型,共同促进领域内的发展,ColabSaprot 集成了 HuggingFace 的模型上传功能,让用户可以无缝将自己训练完的模型上传到模型仓库中供他人使用,在促进社区发展的同时也能提高自身工作的影响力。
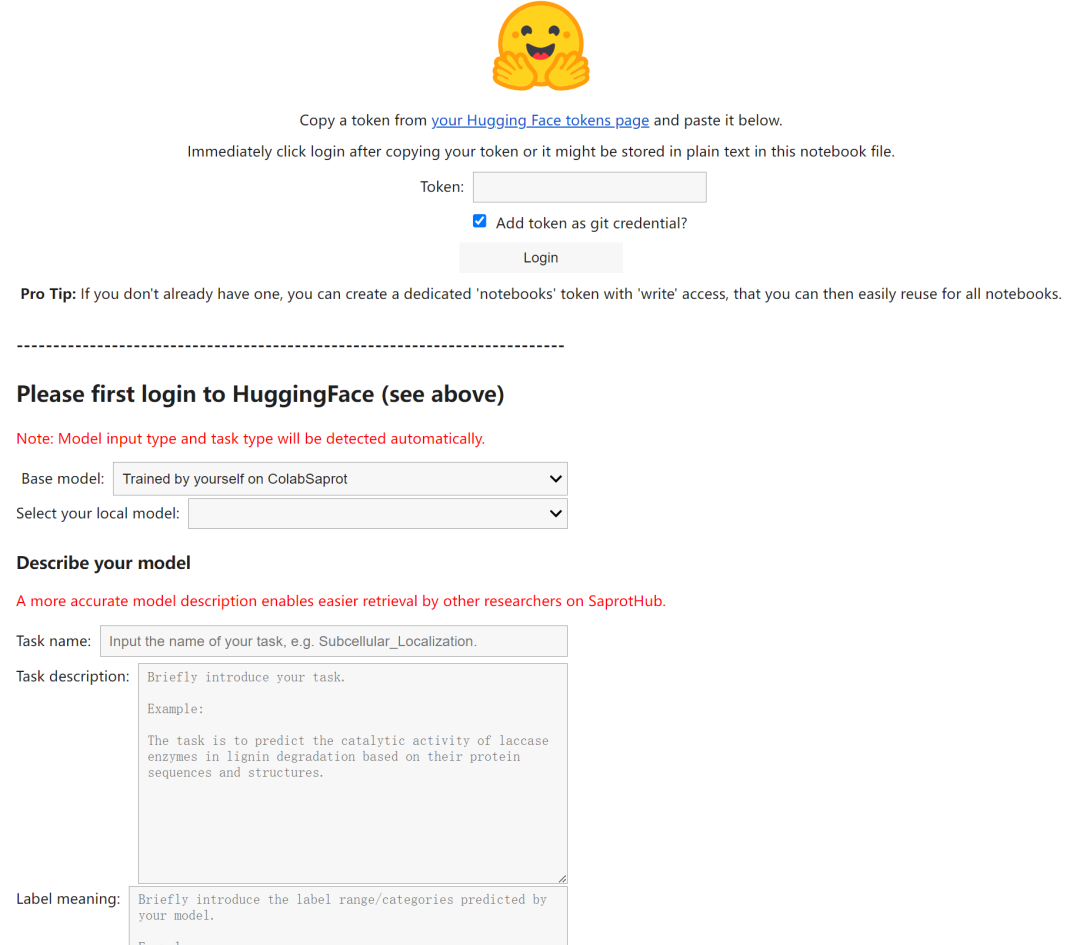
HuggingFace 仓库
作为 SaprotHub 的另一个核心模块,HuggingFace 模型仓库存储了多种已经训练好的模型,可分别用于不同的下游任务预测。
为了方便模型共享和社区协作,ColabSaprot 在训练时采用了 LoRA 的训练机制,通过冻结 backbone 只训练低秩矩阵,大大降低了模型存储和传输的开销。
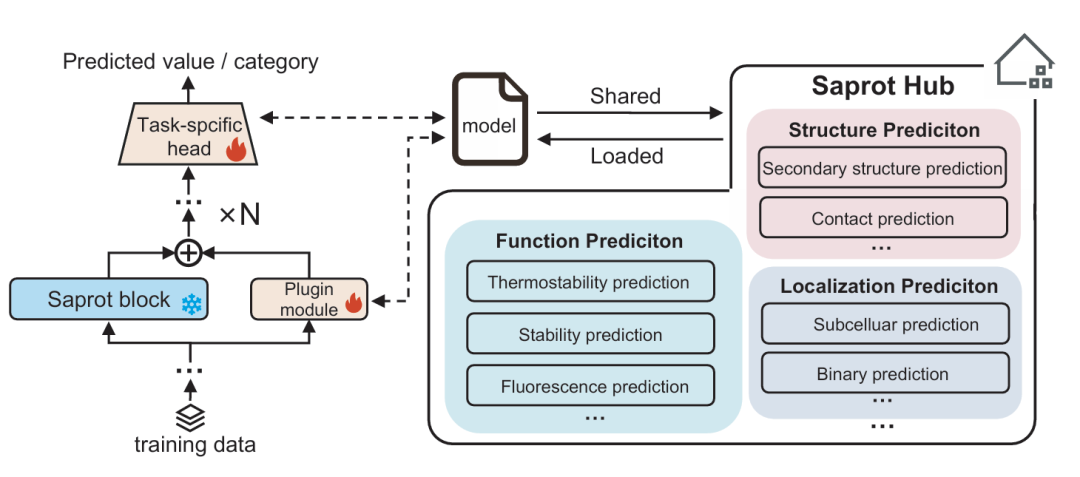
图示:SaprotHub 模型仓库。(来源:论文)
除此之外,为了方便用户在数量众多的模型中快速找到自己感兴趣的模型,作者开发了基于 HuggingFace 的搜索引擎,允许用户通过输入关键词来定位相关的模型以及数据集。
搜索引擎链接:https://huggingface.co/spaces/SaProtHub/SaprotHub-search
实验分析
ColabSaprot 用户测试
为了验证 ColabSaprot 的可用性,作者招募了 12 位没有机器学习相关背景的生物研究者进行了使用测试,结果如下:
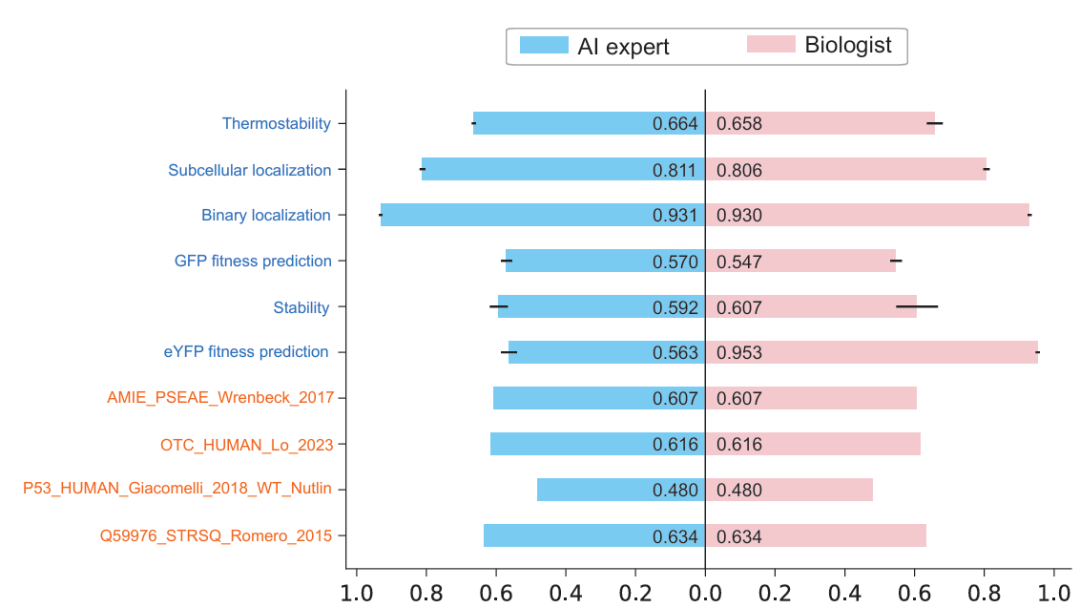
图示:supervised fine-tuning 和zero-shot 预测的结果。(来源:论文)
图中蓝色字体代表监督微调任务,橙色字体代表 zero-shot 突变预测任务。
可以看到,利用 ColabSaprot,即使是没有机器学习背景的用户也能够训练出和 AI 专业的研究者 comparable 的模型,并且用户还能够在 SaprotHub 已有的模型的基础上进一步训练模型,从而缓解了数据不足导致模型性能较弱的情况(见 eYFP fitness prediction)。
而对于突变预测任务,用户可以轻松地获得与编写代码跑出来一致的结果,仅仅需要通过鼠标点击的方式。
湿实验验证
为了验证 ColabSaprot 在实际研究中的可用性,作者对 ColabSaprot 的预测结果进行了多种湿实验验证,包括:
木聚糖酶突变优化(zero-shot)
在预测的 top 20 个突变中,有 13 个突变都使酶活性增强,其中 R59S 和 F212N 分别是原始酶活性的 2.55 倍和 1.88 倍。
TDG 突变优化(zero-shot)
在预测的 top 20 个突变中,有 17 个突变都使编辑效率增强,其中 L74E、H11K 和 L74Q 突变体的编辑效率接近原始蛋白的 2 倍。
GFP 突变优化(supervised fine-tuning)
在通过 GFP 突变数据微调过后,利用模型预测的 top 9 个双点突变中,有 7 个都提高了荧光强度,其中一个突变体达到了野生型 8 倍以上的荧光强度。
结论
研究人员开源的 ColabSaprot 交互式蛋白质语言模型平台,旨在降低 AI 技术的使用门槛,使生物学研究者无需复杂的机器学习背景或编程能力,即可轻松训练和调用 AI 模型。
这一创新举措不仅为研究者提供了强大的工具支持,还显著提升了 AI 技术在蛋白质科学研究中的可及性和实用性。通过 ColabSaprot,更多研究者能够便捷地利用先进 AI 技术,加速研究进程,推动蛋白质科学迈入 AI 赋能的新时代。
论文链接:https://www.biorxiv.org/content/10.1101/2024.05.24.595648v5
使用平台:https://colab.research.google.com/github/westlake-repl/SaprotHub/blob/main/colab/SaprotHub_v2.ipynb
代码仓库:https://colab.research.google.com/github/westlake-repl/SaprotHub/blob/main/colab/SaprotHub_v2.ipynb
好了,本文到此结束,带大家了解了《AI赋能蛋白质研究:SaprotHub让蛋白质AI模型训练和调用不再有门槛!》,希望本文对你有所帮助!关注golang学习网公众号,给大家分享更多科技周边知识!
-
501 收藏
-
501 收藏
-
501 收藏
-
501 收藏
-
501 收藏
-
160 收藏
-
339 收藏
-
212 收藏
-
157 收藏
-
308 收藏
-
416 收藏
-
373 收藏
-
392 收藏
-
464 收藏
-
388 收藏
-
342 收藏
-
238 收藏
-

- 前端进阶之JavaScript设计模式
- 设计模式是开发人员在软件开发过程中面临一般问题时的解决方案,代表了最佳的实践。本课程的主打内容包括JS常见设计模式以及具体应用场景,打造一站式知识长龙服务,适合有JS基础的同学学习。
- 立即学习 543次学习
-

- GO语言核心编程课程
- 本课程采用真实案例,全面具体可落地,从理论到实践,一步一步将GO核心编程技术、编程思想、底层实现融会贯通,使学习者贴近时代脉搏,做IT互联网时代的弄潮儿。
- 立即学习 516次学习
-

- 简单聊聊mysql8与网络通信
- 如有问题加微信:Le-studyg;在课程中,我们将首先介绍MySQL8的新特性,包括性能优化、安全增强、新数据类型等,帮助学生快速熟悉MySQL8的最新功能。接着,我们将深入解析MySQL的网络通信机制,包括协议、连接管理、数据传输等,让
- 立即学习 500次学习
-

- JavaScript正则表达式基础与实战
- 在任何一门编程语言中,正则表达式,都是一项重要的知识,它提供了高效的字符串匹配与捕获机制,可以极大的简化程序设计。
- 立即学习 487次学习
-

- 从零制作响应式网站—Grid布局
- 本系列教程将展示从零制作一个假想的网络科技公司官网,分为导航,轮播,关于我们,成功案例,服务流程,团队介绍,数据部分,公司动态,底部信息等内容区块。网站整体采用CSSGrid布局,支持响应式,有流畅过渡和展现动画。
- 立即学习 485次学习
