LLM时代,计算蛋白质科学进展如何?香港理工大学等发布系统性综述
时间:2025-02-08 12:19:11 355浏览 收藏
目前golang学习网上已经有很多关于科技周边的文章了,自己在初次阅读这些文章中,也见识到了很多学习思路;那么本文《LLM时代,计算蛋白质科学进展如何?香港理工大学等发布系统性综述》,也希望能帮助到大家,如果阅读完后真的对你学习科技周边有帮助,欢迎动动手指,评论留言并分享~
蛋白质语言模型(pLMs)引领计算蛋白质科学发展:一项综述
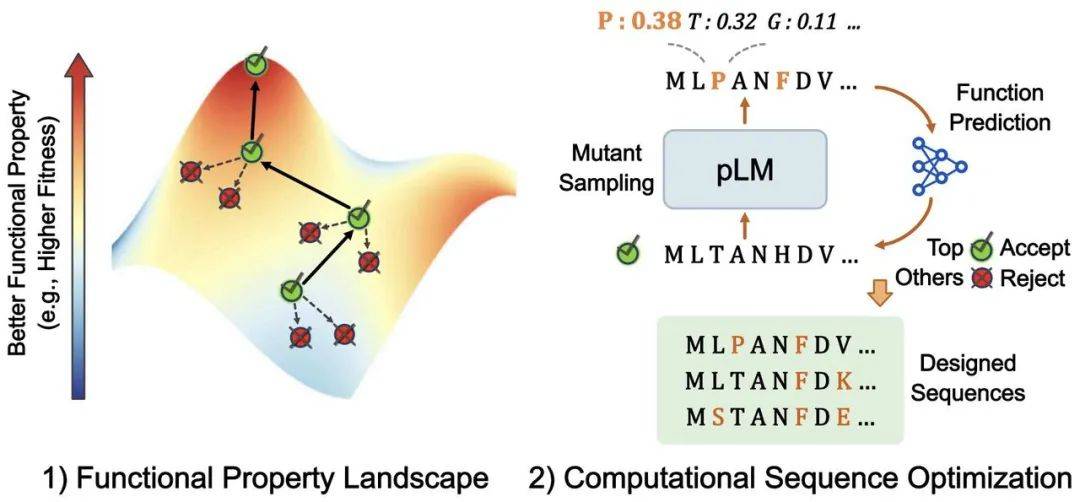
蛋白质是生命的基础,参与几乎所有生命活动。蛋白质的序列、结构和功能之间存在着密切的联系,遵循“序列-结构-功能”范式。 然而,破译蛋白质的“语言”并理解其信息流动规则仍然是巨大的挑战。近年来,大型语言模型(LLMs)技术为计算蛋白质科学带来了新的机遇,催生了蛋白质语言模型(pLMs)。pLMs能够理解氨基酸序列,识别结构和功能信息,并整合多种生物医学数据。
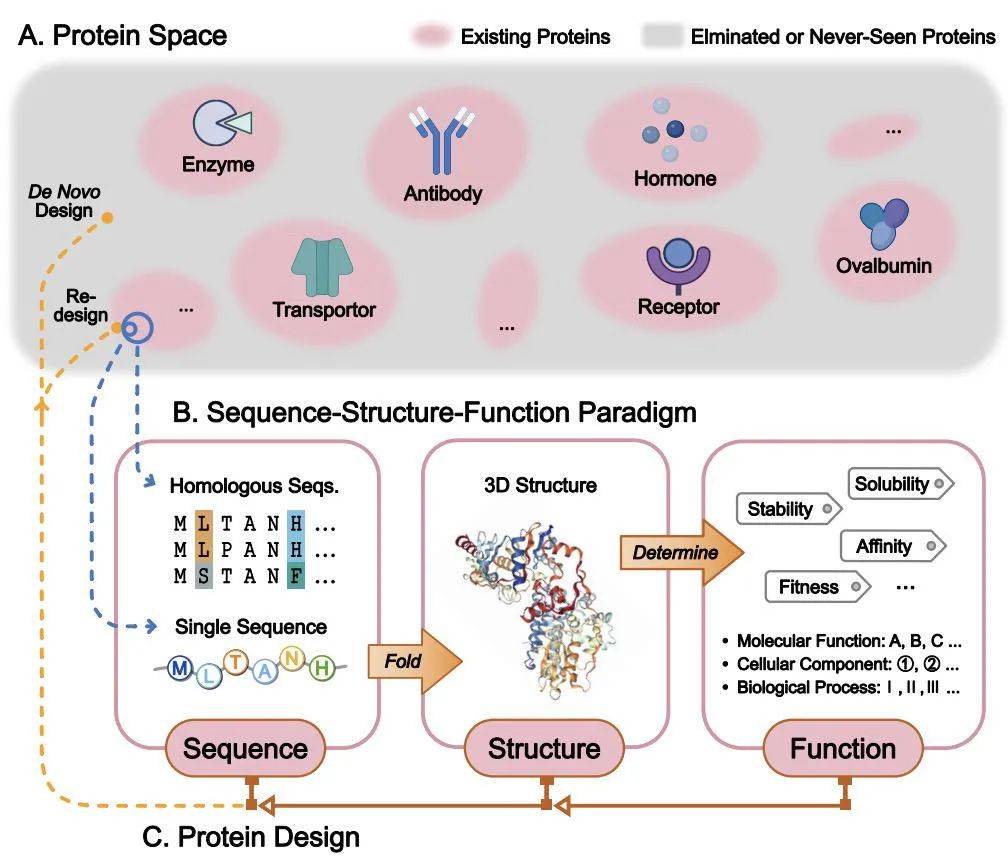
香港理工大学等机构的研究团队近期发表综述文章,系统性地总结了LLM技术在计算蛋白质科学中的应用。该综述首先回顾了蛋白质建模的生物学基础和数据现状,然后对三种类型的pLMs进行了分类和讨论:基于序列的pLMs、结构与功能增强的pLMs以及多模态pLMs。
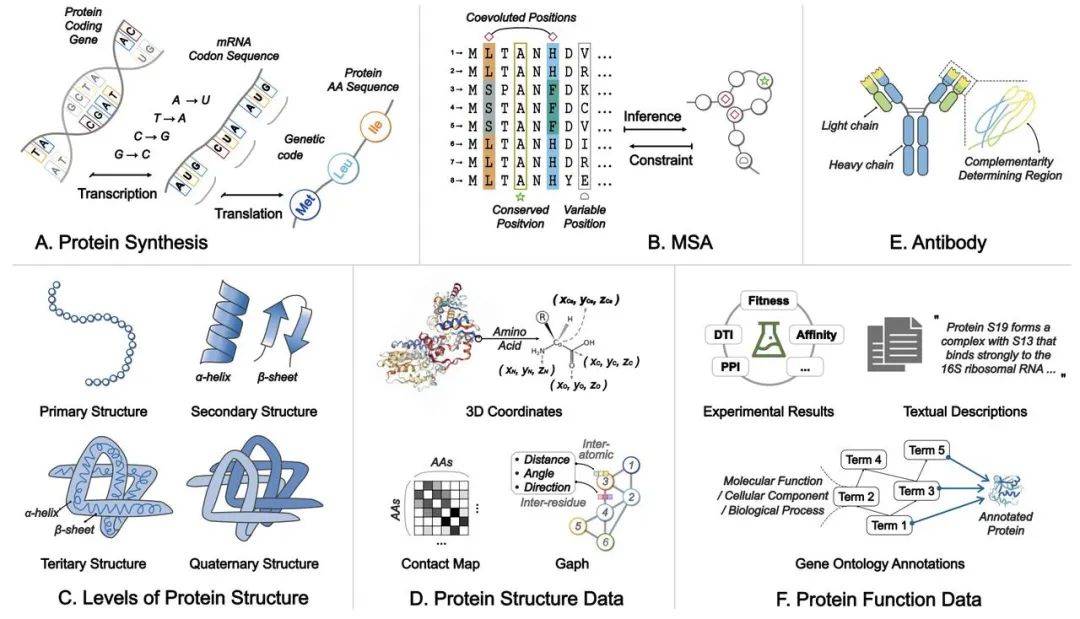
pLMs的类型和应用:
-
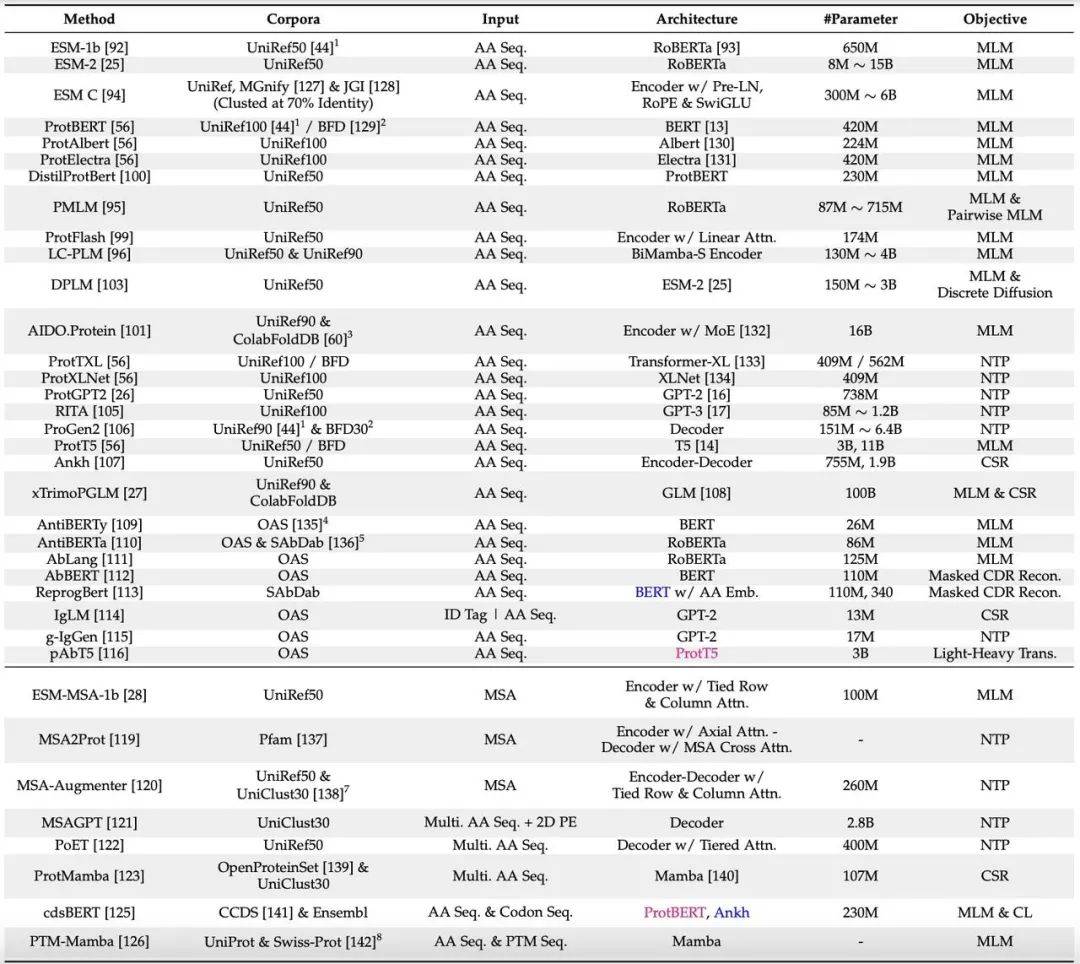
基于序列的pLMs: 这些模型利用氨基酸序列作为输入,学习氨基酸之间的依赖关系,从而推断蛋白质的结构和功能信息。 它们可以进一步细分为基于单序列和基于多序列的模型。
-
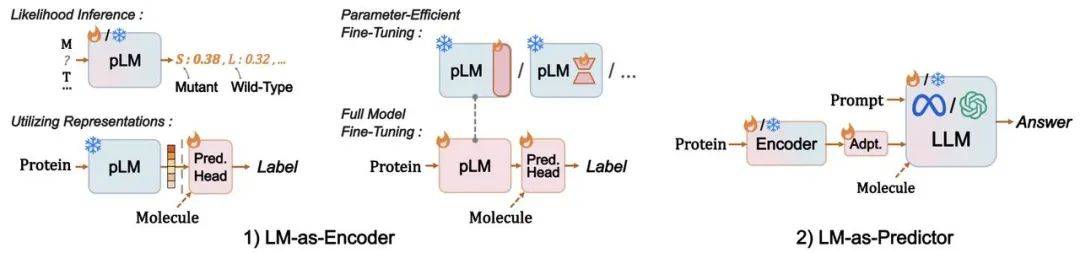
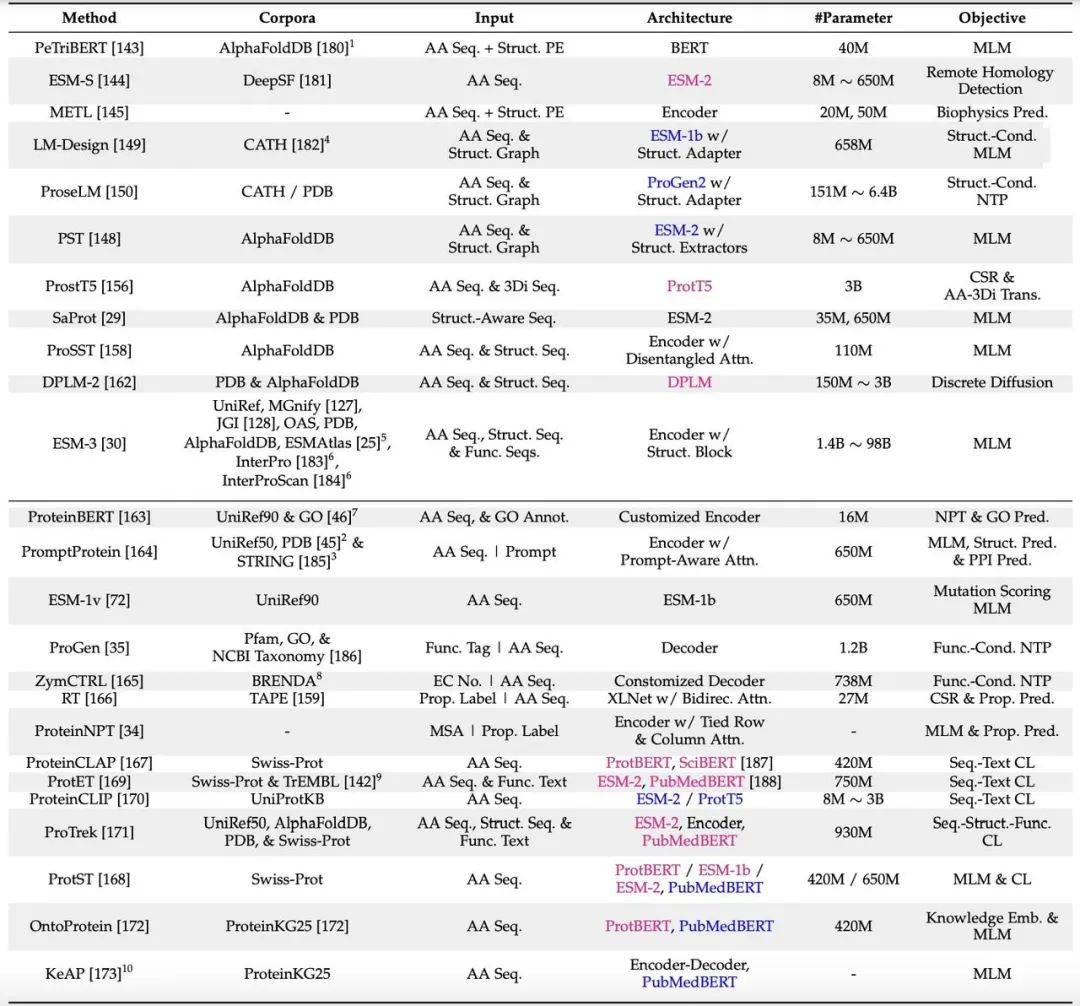
结构与功能增强的pLMs: 这些模型在基于序列的模型基础上,整合了蛋白质的结构和功能信息,从而提高了模型的预测精度和泛化能力。
-
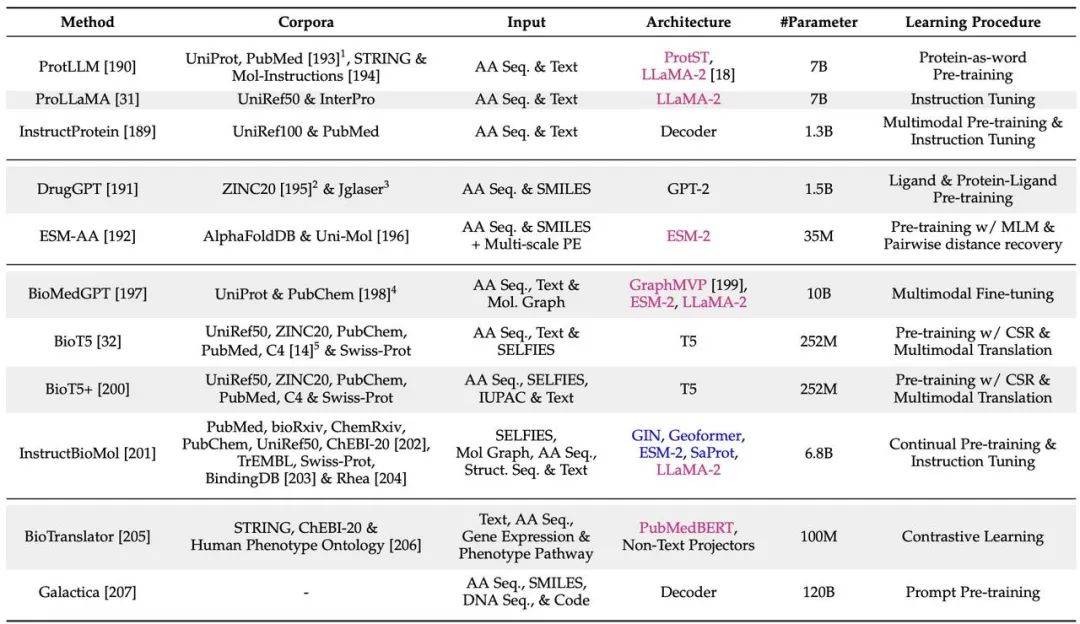
多模态pLMs: 这些模型能够整合多种模态的数据,例如蛋白质序列、结构信息以及描述蛋白质的自然语言文本,从而更全面地理解蛋白质。
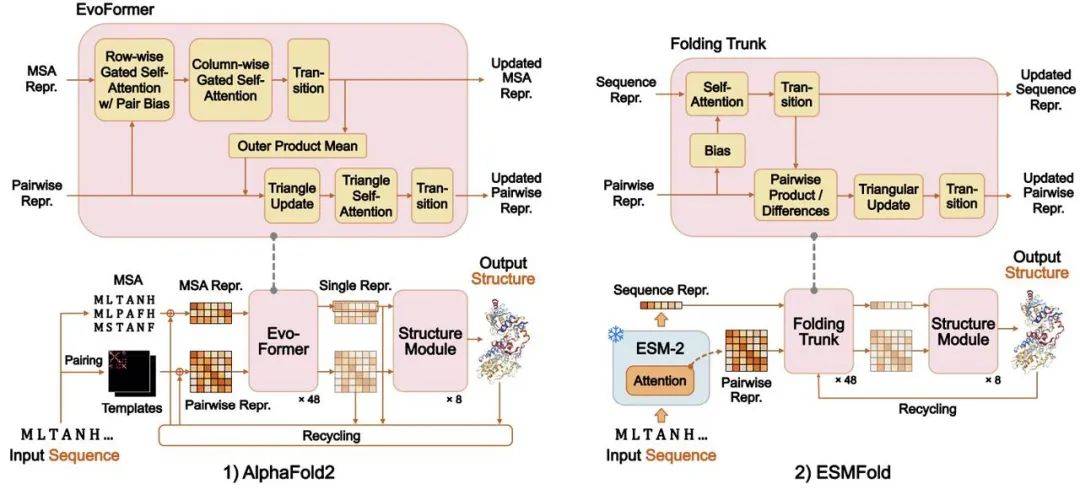
该综述还重点介绍了pLMs在蛋白质结构预测、功能预测和蛋白质设计中的应用,并探讨了其在抗体设计、酶设计和药物靶点发现中的潜力。 例如,pLMs在蛋白质结构预测方面取得了显著进展,例如AlphaFold2和ESMFold等模型已经能够以接近实验精度的水平预测蛋白质结构。
未来展望:
尽管pLMs在计算蛋白质科学领域展现出巨大潜力,但仍面临一些挑战,例如数据稀缺、蛋白质相互作用建模、模型可解释性以及计算效率等问题。 未来的研究方向包括开发更强大的pLMs,改进模型的可解释性,以及将计算方法与实验研究更紧密地结合起来。
该综述为研究人员提供了一个全面了解pLMs在计算蛋白质科学中的应用现状和未来发展方向的宝贵资源。 原文链接:https://arxiv.org/pdf/2501.10282 (请注意,此链接可能需要更新为实际的论文链接)
今天带大家了解了的相关知识,希望对你有所帮助;关于科技周边的技术知识我们会一点点深入介绍,欢迎大家关注golang学习网公众号,一起学习编程~
-
501 收藏
-
501 收藏
-
501 收藏
-
501 收藏
-
501 收藏
-
492 收藏
-
403 收藏
-
484 收藏
-
259 收藏
-
295 收藏
-
338 收藏
-
500 收藏
-
304 收藏
-
298 收藏
-
312 收藏
-
325 收藏
-
417 收藏
-

- 前端进阶之JavaScript设计模式
- 设计模式是开发人员在软件开发过程中面临一般问题时的解决方案,代表了最佳的实践。本课程的主打内容包括JS常见设计模式以及具体应用场景,打造一站式知识长龙服务,适合有JS基础的同学学习。
- 立即学习 543次学习
-

- GO语言核心编程课程
- 本课程采用真实案例,全面具体可落地,从理论到实践,一步一步将GO核心编程技术、编程思想、底层实现融会贯通,使学习者贴近时代脉搏,做IT互联网时代的弄潮儿。
- 立即学习 516次学习
-

- 简单聊聊mysql8与网络通信
- 如有问题加微信:Le-studyg;在课程中,我们将首先介绍MySQL8的新特性,包括性能优化、安全增强、新数据类型等,帮助学生快速熟悉MySQL8的最新功能。接着,我们将深入解析MySQL的网络通信机制,包括协议、连接管理、数据传输等,让
- 立即学习 500次学习
-

- JavaScript正则表达式基础与实战
- 在任何一门编程语言中,正则表达式,都是一项重要的知识,它提供了高效的字符串匹配与捕获机制,可以极大的简化程序设计。
- 立即学习 487次学习
-

- 从零制作响应式网站—Grid布局
- 本系列教程将展示从零制作一个假想的网络科技公司官网,分为导航,轮播,关于我们,成功案例,服务流程,团队介绍,数据部分,公司动态,底部信息等内容区块。网站整体采用CSSGrid布局,支持响应式,有流畅过渡和展现动画。
- 立即学习 485次学习

